30 Jul Ciencia abierta: el poder de la comunicación
La comunicación de resultados científicos sigue unos patrones clásicos bien establecidos desde hace mucho tiempo. Los investigadores que queremos compartir nuestros resultados preparamos un manuscrito y lo enviamos a alguna de las múltiples revistas científicas existentes, las cuales a su vez remiten el manuscrito a otros científicos expertos del mismo campo para su evaluación y comentarios. En el mejor de los casos, tras una o varias rondas de modificaciones, finalmente el manuscrito es publicado. El otro canal de comunicación que habitualmente utilizamos es la presentación de nuestros resultados, incluso antes de su publicación, en reuniones, talleres y congresos. El sistema funciona razonablemente bien para la difusión de nuevos hallazgos, en positivo, particularmente los resultados inesperados, desconocidos, espectaculares, los que abren nuevos horizontes. En cambio, el sistema funciona rematadamente mal para la divulgación de resultados negativos, los que demuestran que algo no funciona como se había publicado anteriormente, o los que van en contra del conocimiento establecido. Raramente estos artículos acaban siendo publicados. Sin embargo todos los investigadores reconocemos el gran valor que tienen los resultados negativos, el saber que algo no funciona, que determinado experimento no debe hacerse de tal o cual manera, pues nos permite evitar malgastar los siempre limitados recursos que tenemos y dedicarlos a otros experimentos con mayores probabilidades de éxito.

Figura 4 del artículo publicado por Gaetan Burgio (ANU) en medium.com donde compartió sus resultados negativos con Argonauta (NgAgo)
La comunicación científica en el siglo XXI ha cambiado. Se ha adaptado a los nuevos canales de comunicación, a las redes sociales (especialmente Twitter, aunque también Facebook y Linkedin), a los blogs, los foros, las listas de correo o los diferentes sistemas que hay hoy en día de intercambio de información de forma inmediata, sin intermediarios, sin cortapisas. Han aparecido diversos canales de comunicación de Ciencia Abierta (Open Science) donde los investigadores comparten libre y voluntariamente sus resultados, los comentan, los critican y, gracias al conocimiento colectivo, se llega muy rápidamente a conclusiones válidas para todos. Y es precisamente en la comunicación de resultados negativos, los que no funcionan como se esperaba, donde los canales de ciencia abierta han triunfado y han encontrado su nicho natural.
Hace pocos días descubrimos, gracias a un proyecto de Ciencia Abierta, que la proteína Argonauta (Ago), proveniente de bacterias, presentada a principios de mayo como el nuevo sistema corta-pega de edición genómica que iba a substituir e incluso mejorar las herramientas CRISPR-Cas, realmente no funcionaba tal y como se había publicado. Gracias a diferentes iniciativas de Ciencia Abierta los investigadores rápidamente empezaron a confirmar los resultados negativos que también habían obtenido con Ago y, en poco tiempo, se comprobó que absolutamente ningún laboratorio había logrado reproducir los fantásticos resultados publicados por un grupo de investigadores chinos en la revista Nature Biotechnology.
La proteína Ago está presente en prácticamente todos los organismos, tanto procariotas (las bacterias) como eucariotas (nosotros), aunque parece realizar diferentes funciones en cada uno de ellos. A principios de 2014, un grupo de microbiólogos holandeses, en colaboración con un microbiólogo español, José Berenguer (CBMSO, Madrid), publicaron un estupendo artículo donde anunciaban que Ago en bacterias funcionaba como una endonucleasa de ADN (es decir, era capaz de cortar la doble hebra del ADN), y que dicha acción la realizaba guiada por pequeñas moléculas de ADN con uno de sus extremos (5’) modificado (fosforilado). Las proteínas Cas de las CRISPR también son endonucleasas de ADN pero sus guías son pequeñas moléculas de ARN y solo son capaces de cortar al lado de secuencias determinadas (PAM). El problema con Ago, inicialmente descubierta en una bacteria, Thermus thermophilus, que vive en entornos extremos, con altas temperaturas, como las fumarolas hidrotermales, es que está adaptada a funcionar a temperaturas elevadas (>65ºC) mientras que la temperatura fisiológica de nuestro cuerpo y nuestras células es sensiblemente inferior (37ºC), lo cual parecía impedir su funcionamiento normal en células animales.
En mayo de este año, el laboratorio del Dr. Chunyu Han (Hebei University of Science and Technology, Shijiazhuang, Hebei, China) pareció haber dado con la solución al problema, al aislar Ago de una haloarquea (que vive en ambientes salinos) denominada Natronobacterium gregoryi, de ahí que la proteína fuera rebautizada como NgAgo. Los investigadores chinos demostraron en su artículo que NgAgo actuaba como un verdadero editor génico en células humanas en cultivo. También comprobaron que NgAgo no necesitaba PAM (una de las limitaciones de las CRISPR-Cas) ni parecía cortar de forma relevante en secuencias parecidas pero no idénticas a las presentes en la guía de ADN (otra diferencia importante con las CRISPR-Cas, cuya capacidad de cortar en secuencias parecidas del genoma, los denominados off-targets, está limitando y retrasando su utilización inmediata en terapia génica). Se trataba pues de la quintaesencia de los editores genómicos, la cuarta generación (tras ZFNs, TALENs y CRISPRs), que mejoraba sobre el papel a la anterior (CRISPR-Cas).
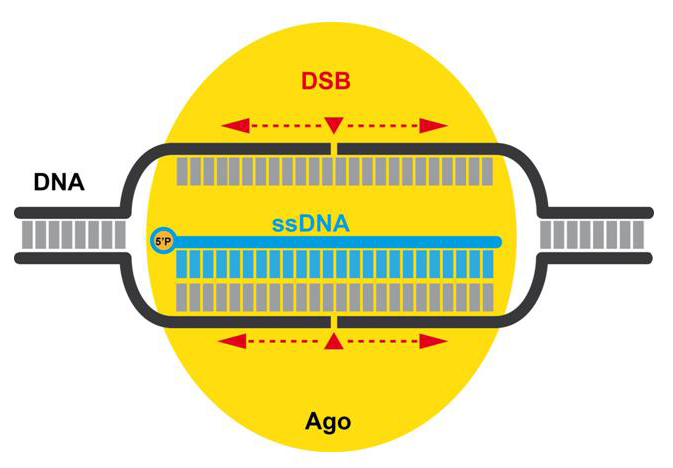
Mecanismo de acción propuesto para la proteína Argonauta como editor genómico (esquema realizado por Lluís Montoliu)
NgAgo se empezó a distribuir gracias a repositorios públicos de reactivos (Addgene) a principios de junio y, desde el mismo día de su lanzamiento, centenares sino miles de laboratorios de todo el mundo la solicitaron para empezar a usarla en sus proyectos. Sin embargo, pocas semanas después empezaron los rumores, los comentarios por email, por WhatsApp, en los congresos y reuniones, dentro de las sociedades científicas, y finalmente, también en las diferentes iniciativas de Ciencia Abierta, en los grupos de discusión abierta de Google, en Twitter, en los blogs… Algo no iba bien. NgAgo no parecía hacer lo que supuestamente debía de hacer. Ningún laboratorio parecía ser capaz de reproducir los resultados publicados, ni se podía confirmar actividad de NgAgo como editor génico en ratones, ratas o peces cebra, entre las especies modelo de laboratorio investigadas.
Gaétan Burgio, un genetista francés que trabaja en The John Curtis School of Medical Research de la Australian National University en Canberra, experto en el uso de modelos animales para el estudio de la malaria y otras enfermedades infecciosas, fue uno de los laboratorios que solicitó NgAgo y también uno de los que vio frustradas sus expectativas al ser incapaz de reproducir los resultados publicados. Sin embargo, Gaétan Burgio, a diferencia de otros, decidió compartir sus resultados negativos en un proyecto de Ciencia Abierta, fuera de los canales tradicionales, los mostró abiertamente y los interpretó de la manera más objetiva que supo, concluyendo que NgAgo no tenía actividad como editor genético. Dicha publicación generó un tsunami de respuestas desde todos los lugares del mundo, de otros investigadores que confirmaban que tampoco a ellos les había funcionado NgAgo, lo cual permitió concluir, en pocas horas, que el anunciado nuevo sistema NgAgo no funcionaba tal y como se había publicado.
Este ejemplo de Ciencia Abierta, además de ilustrar la generosidad y espíritu colaborativo de Gaétan Burgio, demuestra cuán importante es conocer que un reactivo no funciona en Ciencia cuanto antes. En apenas dos meses desde la publicación inicial hemos confirmado que NgAgo no funciona tal y como se describió (y ahora habrá que averiguar por qué, aunque este es ya otro tema que puede tener diferentes explicaciones) y esto ha permitido a muchos laboratorios detener y terminar experimentos que tenían planeados o en marcha, experimentos en los que se iban a invertir cantidades ingentes de recursos que habrían sido desgraciadamente malgastados.
Gracias a la Ciencia Abierta hemos podido evitar gastar millones de euros, evitar usar miles de animales y evitar perder el tiempo en un reactivo que, ahora sabemos, no funciona tal y como se describió. Esperemos que pronto sepamos qué ha sucedido con NgAgo y cuál es la razón de la falta de reproducibilidad de los resultados originales. Estoy seguro que, de nuevo, a través de algún canal de Ciencia Abierta, sabremos la respuesta más pronto que tarde. Sigamos atentos!



